Sagemaker(チュートリアル Processing編)
概要
公式チュートリアルを使って、SageMaker Processingの理解をしてみる。
Processingに関する 公式ブログによると、前処理や後処理を独立したジョブとして実行できる機能。
Processingを使う際の、考えられるメリットとしては以下。
- 前処理などはnotebookのインスタンスリソースでは不足しがちなので
これを別のマネージドなリソースで実行できると嬉しい。 - 前処理、学習、後処理、評価、などを疎に作り再利用もできる。(これが嬉しいかどうかは微妙かと思っており、詳しくは後述)
チュートリアルについて
概要
SKLearnProcessorを使うチュートリアルと、
ScriptProcessorを使うチュートリアルの2パターンが用意されている。
両者は、前処理などを実行する際に使用するdockerイメージが違う。SKLearnProcessorはscikit-learnをimportできるようあらかじめsklearnがinstallされたdockerイメージが使われ、ScriptProcessorは独自のdockerイメージを使う。
そのため、前処理などにscikit-learnしか使わない場合は前者を使えばいいし、それ以外なら後者、という棲み分け。
SKLearnProcessorは以下のように使う。
from sagemaker.sklearn.processing import SKLearnProcessor
sklearn_processor = SKLearnProcessor(framework_version='0.20.0',
role=role,
instance_type='ml.m5.xlarge',
instance_count=1)
ScriptProcessorは以下のような感じ。
from sagemaker.processing import ScriptProcessor
spark_processor = ScriptProcessor(base_job_name='spark-preprocessor',
image_uri=spark_repository_uri,
command=['/opt/program/submit'],
role=role,
instance_count=2,
instance_type='ml.r5.xlarge',
max_runtime_in_seconds=1200,
env={'mode': 'python'})
ScriptProcessorの方はdockerのイメージURIなどを指定しているのに対し、SKLearnProcessorの方はバージョンなどしか指定していない。SKLearnProcessorはAWSが提供しているsklearn用のdockerイメージが勝手に使われるのだろう。
以降、気になるポイントやつまづいたところなどだけピックアップして記載する。
コピペしても動かないので、詳しくは
公式チュートリアルを参照のこと。
SKLearnProcessor
scikit-learnを使えるコンテナ内でProcessingジョブが実行される。
s3fsモジュールのインストール
S3からデータを読み込む以下のところで、いきなりエラーになる。
import pandas as pd
input_data = 's3://sagemaker-sample-data-{}/processing/census/census-income.csv'.format(region)
df = pd.read_csv(input_data, nrows=10)
df.head(n=10)
s3fsというモジュールが足りないので、適当なセルで!pip install s3fsしておくこと。
もっとも、ここの処理は単にデータをheadしているだけなので、実際の前処理ジョブには必要ない。(前処理ジョブが実行されるdockerイメージ内ではすでにs3fsがインストールされている)
前処理ジョブ用のスクリプトを作成
preprocessing.pyというスクリプトを作成するセルがある。(%%writefile preprocessing.pyと書いてあるセル)
単にmain関数内に前処理をつらつら書いているスクリプト。
可変値はargparseでコマンド引数を定義しておいて、SKLearnProcessorでそれを指定してあげる、という流れになる。
それらのコマンド引数はrunメソッドのargumentsにlistで渡してやれば良い。
チュートリアルのコードでは下記のようにargumentsを指定している。
sklearn_processor.run(code='preprocessing.py', # 前処理ジョブで実行するスクリプト
inputs=[ProcessingInput(
source=input_data,
destination='/opt/ml/processing/input')],
outputs=[ProcessingOutput(output_name='train_data',
source='/opt/ml/processing/train'),
ProcessingOutput(output_name='test_data',
source='/opt/ml/processing/test')],
arguments=['--train-test-split-ratio', '0.2'] # ここでpreprocessing.pyのコマンド引数を指定
)
入出力ファイルの指定
Processingジョブ内で扱う入出力ファイルは、基本的にS3=>ジョブコンテナ内のファイルシステム=>S3という流れで格納される。
ジョブ実行中はコンテナ内のストレージで処理し、実行前後はS3に置く、ということである。
入出力ファイルのS3 URIとコンテナ内のファイルパスは、上記runメソッドのinputs, outputsで紐付けてやる必要がある。(ファイルコピーは勝手に実行される)
一例としては下図のような流れになる。
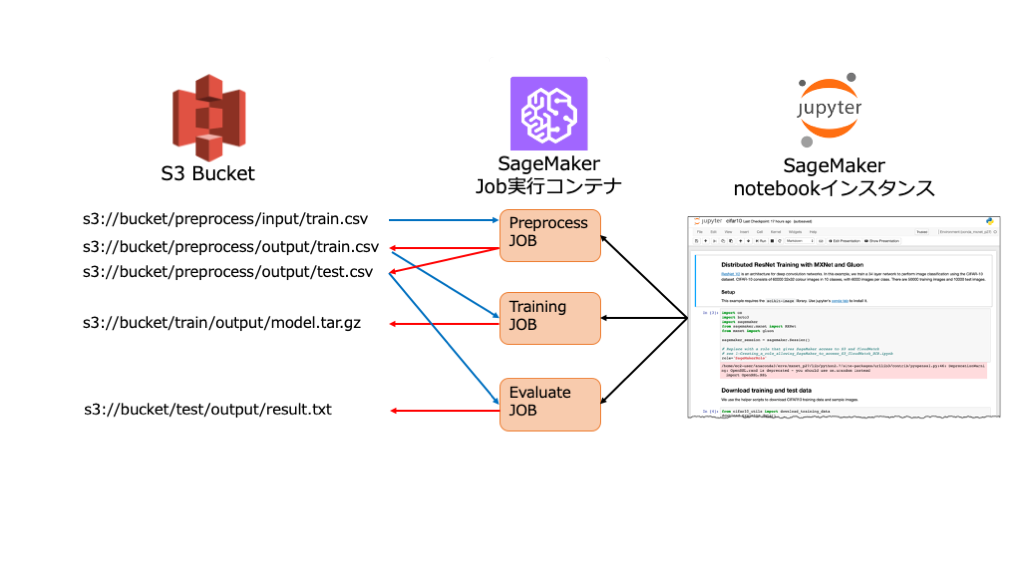
ジョブは一時的なJob実行コンテナで起動するため、入出力ファイルの永続化には基本的にS3を使おう、ということ。
Processingジョブの一覧取得
参考: [小ネタ]Amazon SageMaker Processingのジョブの一覧をBoto3を使って確認する
学習や推論のジョブは、AWSコンソールのSageMakerからブラウザ上で確認できるのだが、
なぜかProcessingジョブはコンソール上から確認できない仕様になっているらしい。
そのためboto3を使ってProcessingジョブのlistを取得する必要がある。
import boto3
import pandas as pd
sm = boto3.Session().client('sagemaker')
jobs = sm.list_processing_jobs()
pd.DataFrame(jobs['ProcessingJobSummaries'])

取得するジョブのフィルタリングや、ジョブ詳細を確認するオプションもある。
詳しくは上記の参考リンクを確認すること。
ScriptProcessor
独自にbuildしたdockerイメージから作られるコンテナ内でProcessingジョブが実行される。
基本的なジョブ発行手順は上記SKLearnProcessorの例と同じ。
あらかじめ独自の前処理用dockerイメージをECRにpushしておき
そのイメージURIを指定して実行する。
下記、再掲。image_uriを指定してあげることで任意のコンテナに含まれる前処理コマンドを実行するジョブを発行できる。
from sagemaker.processing import ScriptProcessor
spark_processor = ScriptProcessor(base_job_name='spark-preprocessor',
image_uri=spark_repository_uri,
command=['/opt/program/submit'],
role=role,
instance_count=2,
instance_type='ml.r5.xlarge',
max_runtime_in_seconds=1200,
env={'mode': 'python'})
SKLearnProcessorのチュートリアルnotebookの末尾に「Running processing jobs with your own dependencies」というのが独自のProcessingジョブ実行手順。
Dockerfile作って
%%writefile docker/Dockerfile
FROM python:3.7-slim-buster
RUN pip3 install pandas==0.25.3 scikit-learn==0.21.3
ENV PYTHONUNBUFFERED=TRUE
ENTRYPOINT ["python3"]
ECRにpushして
import boto3
account_id = boto3.client('sts').get_caller_identity().get('Account')
ecr_repository = 'sagemaker-processing-container'
tag = ':latest'
uri_suffix = 'amazonaws.com'
if region in ['cn-north-1', 'cn-northwest-1']:
uri_suffix = 'amazonaws.com.cn'
processing_repository_uri = '{}.dkr.ecr.{}.{}/{}'.format(account_id, region, uri_suffix, ecr_repository + tag)
# Create ECR repository and push docker image
!docker build -t $ecr_repository docker
!$(aws ecr get-login --region $region --registry-ids $account_id --no-include-email)
!aws ecr create-repository --repository-name $ecr_repository
!docker tag {ecr_repository + tag} $processing_repository_uri
!docker push $processing_repository_uri
ScriptProcessorオブジェクトを生成して
from sagemaker.processing import ScriptProcessor
script_processor = ScriptProcessor(command=['python3'],
image_uri=processing_repository_uri,
role=role,
instance_count=1,
instance_type='ml.m5.xlarge')
runする。(超手抜き)
script_processor.run(code='preprocessing.py',
inputs=[ProcessingInput(
source=input_data,
destination='/opt/ml/processing/input')],
outputs=[ProcessingOutput(output_name='train_data',
source='/opt/ml/processing/train'),
ProcessingOutput(output_name='test_data',
source='/opt/ml/processing/test')],
arguments=['--train-test-split-ratio', '0.2']
)
script_processor_job_description = script_processor.jobs[-1].describe()
Dockerのbuildと、ECRへのpushをnotebook上でやるのが若干ダルいしnotebookの再実行もしづらいので、これらは流石に別のターミナル上でやっておいた方が良いと思う。
所感
そもそも前処理、学習、評価などのジョブを分ける前提として
ある程度各処理が作り込まれていないと、かえって面倒な気がする。
つまり、各処理を試行錯誤している段階ではライトにトライ&エラーを繰り返したいので
わざわざジョブに括りだして実行するのは大げさか。
これらジョブ分離の恩恵が得られるのは、
例えばデイリーのデータでモデルを更新し継続的に評価したいとき、など
ある程度作り込みが終わった後かもしれない。前処理と学習を疎に作れる、というメリットはあるか?
この学習モデルを推論APIにデプロイしたときに、同じ前処理を実行するように
しないといけない。結局、前処理ジョブ用のコンテナとして括りだした処理を
推論API用のイメージにも含めないといけないので、ソースコードの管理は慎重に行う必要がある。
(例えば2重管理にはならないように気をつけたい)